Sistema de Identificação de Microorganismos AXIMA - Características
Apenas Três Etapas do Preparo da Amostra até a Identificação
Preparo de Amostra
Prepare uma colônia de microrganismos (ou cultura em caldo).
Não é necessário nenhum teste de coloração de Gram ou avaliação morfológica prreviamente. Não é preciso ter experiência especializada ou conhecimento anterior sobre microrganismos. A análise pode ser realizada com apenas alguns microgramas de amostra, contendo cerca de 105 bactérias. A presença de múltiplas estirpes bacterianas em colônias no meio ágar não representa um obstáculo para a análise.
Passo 1
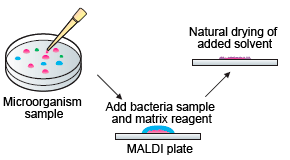
Aplique a amostra de bactérias coletada e o reagente de matriz em uma placa MALDI especial.
O método básico de preparo da amostra é extremamente simples: basta misturar a amostra de bactérias e o reagente de matriz na placa MALDI*1.
As placas MALDI descartáveis simplificam o manuseio. Você pode escolher entre os métodos de preparo de amostras ideais pré-existentes (protocolos) de acordo com suas necessidades.
Passo 2
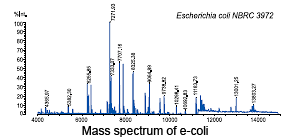
Meça usando MALDI-TOF MS. O espectro de massa único para as espécies bacterianas é obtido rapidamente.
As medições são realizadas automaticamente, usando métodos existentes. Nenhuma experiência em espectrometria de massa é necessária. Não é necessário verificar cada espectro de massa..
Passo 3
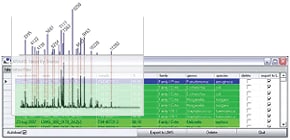
Compare o espectro de massa adquirido com os bancos de dados de espectro registrados no software de análise para classificar e identificar o microorganismo.
Toda a sequência de operações, desde a pesquisa no banco de dados até a exibição dos resultados da identificação, é automatizada. Nenhum ajuste de parâmetro é necessário.
Bancos de Dados Confiáveis
Os bancos de dados confiáveis fornecem um suporte poderoso para identificação de microrganismos.
Banco de Dados de Microorganismos Abrangente e Incomparável
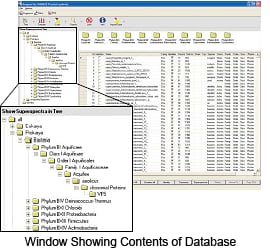
Banco de dados de espectro totalmente validado
SuperSpectraTM Número de espectros de massa registrados*2: 2.000 min.
O SuperSpectraTM contém grupos de pico que podem atuar como marcadores para oferecer detecção estável de espécies bacterianas comumente detectadas sob várias condições. Esses grupos de picos marcadores são extraídos de pelo menos 15 espectros de massa para cada cepa de microrganismo que são obtidos em vários laboratórios sob muitas condições de medição. Produz resultados de identificação confiáveis na maioria das condições.
A identificação de microorganismos por MALDI-TOF MS anteriormente sofria de problemas com a reprodutibilidade do espectro de massa. No entanto, o SuperSpectraTM supera esses problemas para reduzir significativamente os erros durante a identificação dos microorganismos. O SuperSpectraTM e outros bancos de dados contêm aproximadamente 40.000 espectros de massa. Bancos de dados podem ser fornecidos mesmo para espécies bacterianas raramente detectadas.
Personalização do Banco de Dados para Suportar todas as Áreas
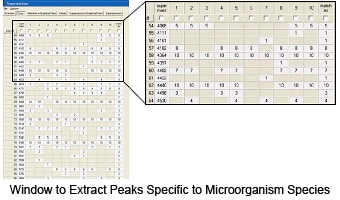
É fácil registrar um novo microrganismo no banco de dados. As operações para extrair os grupos de pico que podem atuar como marcadores de vários espectros de massa são simples e intuitivas. Isso permite que o usuário desenvolva rapidamente um banco de dados de alta qualidade.
*1 Sempre inative amostras infecciosas ou possivelmente infecciosas antes do manuseio.
*2 O número total de espectros de massa pode mudar devido à otimização do banco de dados.
Fornece Melhor Capacidade de Discriminação
Strain Solution (opcional)
Caracterização Bacteriana Altamente Precisa, Rápida e Simples Usando MALDI-TOF MS
O software Strain Solution usa um espectrômetro de massa MALDI-TOF, baseado no método S10-GERMS, para discriminar bactérias em nível de subespécie ou cepa.
As bactérias podem ser caracterizadas por marcadores pré-registrados que permitem ao usuário diferenciar as subespécies e cepas de bactérias. Posteriormente, esses marcadores são combinados com os dados de medição MALDI-TOF MS para determinar a cepa de bactéria alvo.
Características
- Obtenha resultados de discriminação mais precisos e confiáveis do que os métodos convencionais de identificação de microorganismos usando impressão digital MALDI-TOF-MS.
- Caracterize rapidamente bactérias por diferenças genéticas na sequência de bases usando MALDI-TOF MS.
- Crie e personalize bancos de dados de cepas bacterianas.
- Realize análises filogenéticas moleculares usando software externo.
Aplicações
O software amplia a gama de aplicações possíveis usando o Sistema de Identificação de Microorganismos AXIMA-iD Plus, permitindo a caracterização bacteriana até o nível da cepa. Este software é ideal para:
- Gestão da coleção de cultura bacteriana
- Discriminação de bactérias geneticamente semelhantes que são difíceis de diferenciar pelo sequenciamento do gene 16S rRNA
- Análise filogenética molecular
Método S10-GERMS*
O método S10-GERMS vincula informações de massa molecular de proteínas ribossomais obtidas usando MALDI-TOF MS a informações genéticas para classificar e identificar microorganismos. O foco no operon S10-spc-alfa, para o qual cerca de metade das proteínas ribossomais são codificadas, permite que a informação genética seja associada à informação de massa molecular de MALDI-TOF MS, mesmo para espécies bacterianas não sequenciadas pelo genoma.
* S10-GERMS: S10-spc-alpha operon Espectro de massa da proteína ribossomal codificada pelo gene
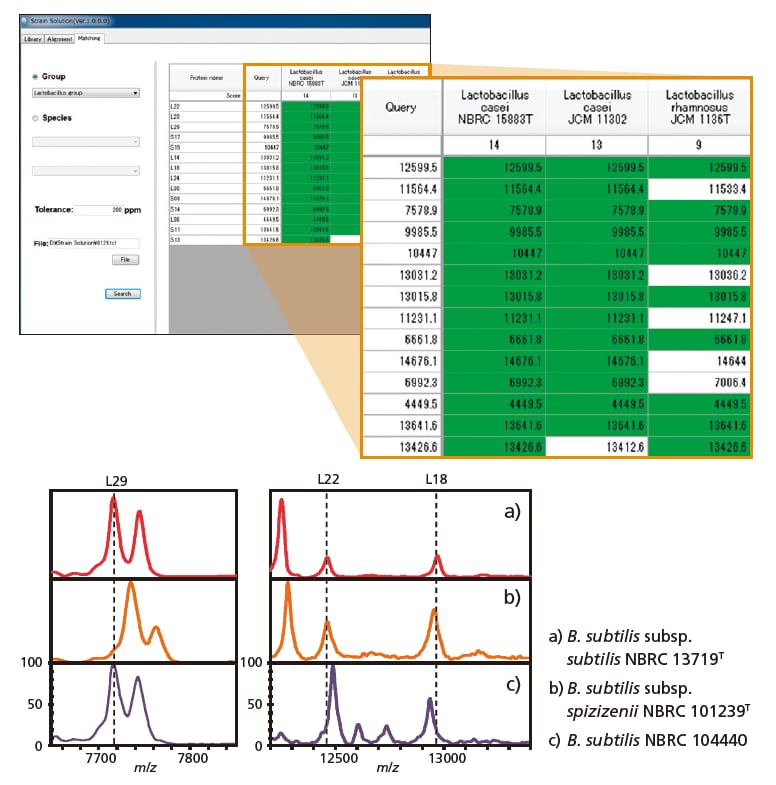
Figure: Example of Discriminating Bacterial Strains with 99.9 % 16S rRNA Gene Homology Using the S10-GERMS Method
Fluxo de Trabalho usando o Software Strain Solution
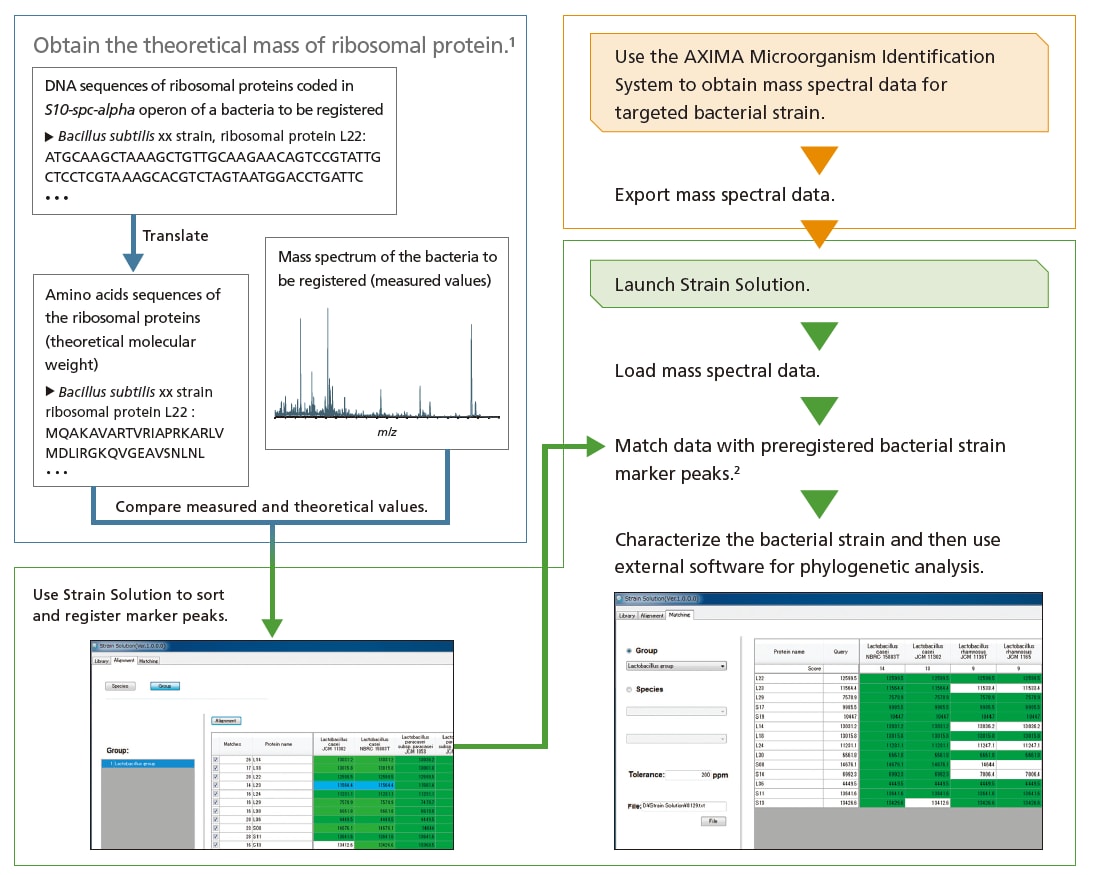
1. As porções indicadas em “obter a massa teórica da proteína ribossomal” indicam funcionalidade não incluída no software.
2. O software não inclui picos de marcadores registrados para amostras diferentes da amostra de teste. Os picos de marcadores de cepas bacterianas para as espécies bacterianas alvo devem ser previamente registrados.


